R语言数学建模(二)—— tidymodels
文章目录
前言
上一节内容中,简单介绍了使用R语言对数据进行建模的一个工作流程。在R中,为了方便进行数学建模,有一个实用的包——tidymodels。这一节将通过学习这个包,体会其进行R语言建模的流程。
一、示例数据集
在上一节就讲到过,想要进行R建模,首先需要对数据有一定的了解。本节将通过一个实例来进行建模学习。让我们首先来认识一下这个示例数据集:Ames住房数据集。
library(modeldata) # tidymodels包也加载此包
data(ames)
# 或者这样加载
data(ames, package = "modeldata")
dim(ames)
#> [1] 2930 74
可以看到ames是一个2930行74列的数据集。该数据集包含的房产信息包括以下内容的列:
-
房屋特征(bedrooms,garage,fireplace,pool,porch,etc.)
-
位置(社区)
-
地段信息
-
状况质量评级
-
销售价格
我们本次建模的目标是根据我们所拥有的其他信息预测房屋的销售价格。让我们先来观察一下房子销售价格的分布:
library(tidymodels)
#> ── Attaching packages ────────────────────────────────────── tidymodels 1.1.1 ──
#> ✔ broom 1.0.5 ✔ rsample 1.2.0
#> ✔ dials 1.2.0 ✔ tibble 3.2.1
#> ✔ dplyr 1.1.3 ✔ tidyr 1.3.0
#> ✔ ggplot2 3.4.4 ✔ tune 1.1.2
#> ✔ infer 1.0.5 ✔ workflows 1.1.3
#> ✔ parsnip 1.1.1 ✔ workflowsets 1.0.1
#> ✔ purrr 1.0.2 ✔ yardstick 1.2.0
#> ✔ recipes 1.0.8
#> ── Conflicts ───────────────────────────────────────── tidymodels_conflicts() ──
#> ✖ purrr::discard() masks scales::discard()
#> ✖ dplyr::filter() masks stats::filter()
#> ✖ dplyr::lag() masks stats::lag()
#> ✖ recipes::step() masks stats::step()
#> • Learn how to get started at https://www.tidymodels.org/start/
tidymodels_prefer()
ggplot(ames, aes(x = Sale_Price)) +
geom_histogram(bins = 50, col = "white")
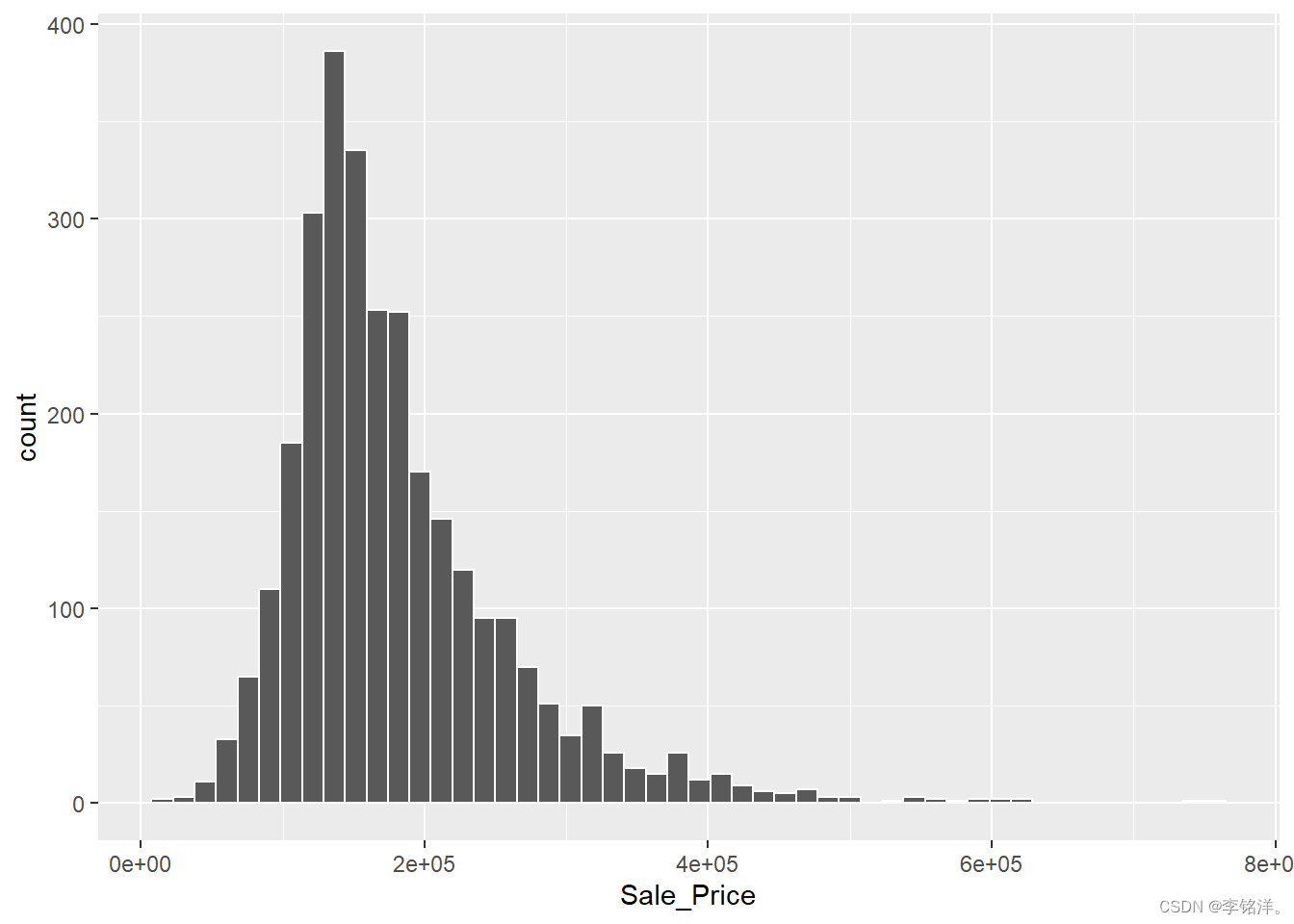
曲线图可以看出,数据不对称,便宜的房子比昂贵的房子要多。处理这样的数据可以考虑使用对数变换。这种变换的好处在于,不会以负销售价格来预测任何房屋,且预测昂贵房屋的错误不会对模型产生影响。统计学的角度来讲,对数变换也可能以一种使推断更合理的方式稳定方差。我们可以使用类似步骤可视化转换后的数据:
ggplot(ames, aes(x = Sale_Price)) +
geom_histogram(bins = 50, col = "white") +
scale_x_log10()
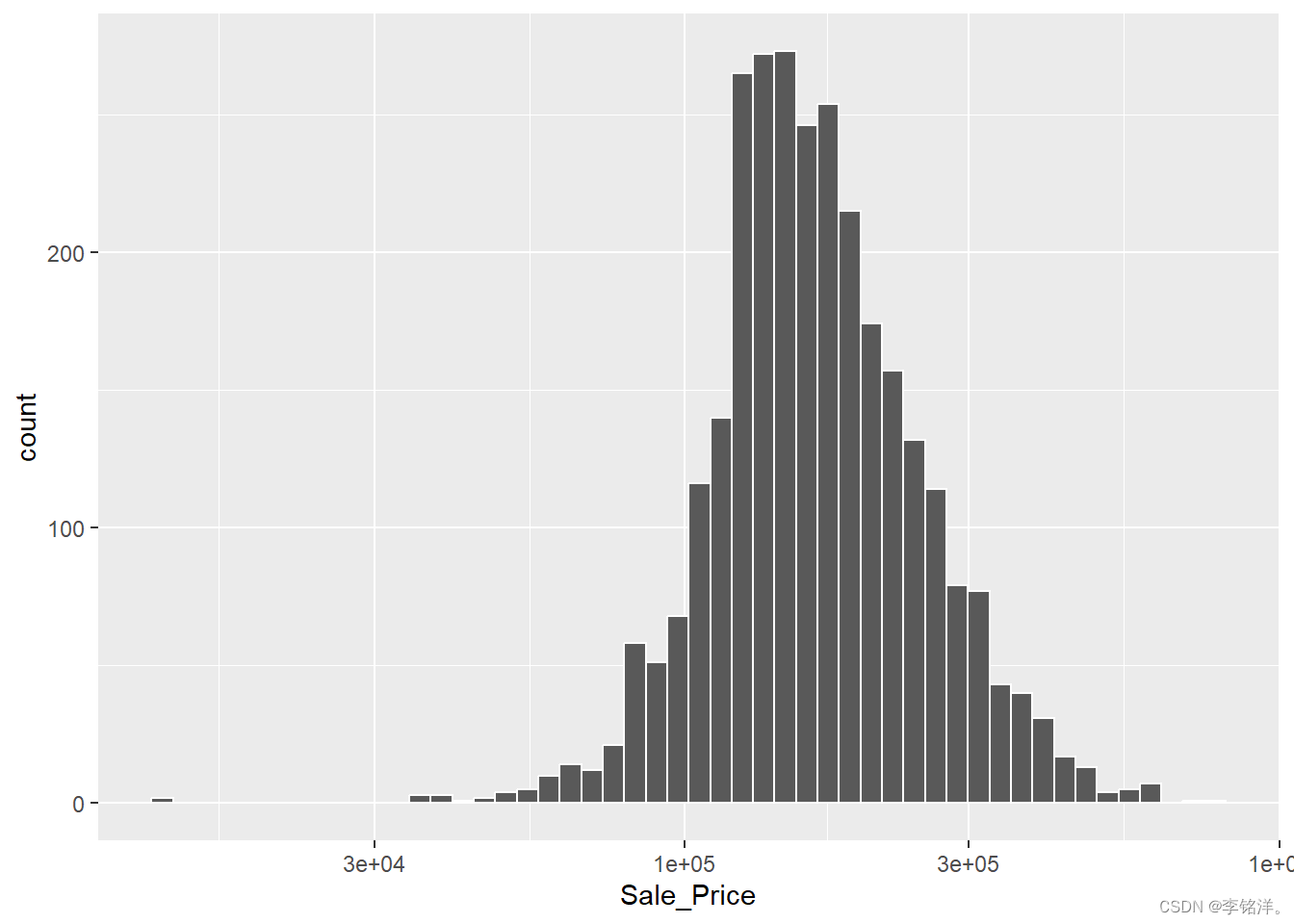
转换结果的缺点主要涉及对模型结果的解释。另外对于转换后的模型系数的单位可能更难解释。例如,均方根误差(RMSE)是回归模型中常用的性能指标。计算中使用观测值与预测值间的差值。如果销售价格在对数标尺上,这些差额(残差)也在对数标尺上,这样的尺度上,RMSE为0.15的模型的质量将很难理解。
尽管如此,这里我们使用的模型使用了对数转换的结果,现在,将其进行对数转换:
ames <- ames |> mutate(Sale_Price = log10(Sale_Price))
在任何建模开始前,探索数据是至关重要的。这些住房数据的特点对如何处理和建模数据提出了有趣的挑战。在这一阶段可以探索的问题包括:
-
单个预测因子的分布有没有什么值得注意的地方?是否有很大的偏斜?
-
预测因子间是否有很强的相关性?有些相关因素是多余的吗?
-
预测因子与结果之间是否存在关联?
二、拆分数据集
创建有用的模型有几个步骤,包括参数估计、模型选择和调整以及性能评估。新项目开始前,通常会有一个初始的有限数据集可以用于不同的步骤或任务。建模时,如何使用数据是一个要首要考虑的因素。
当数据量很大时,明智的策略是为不同的任务分配特定的数据子集,而不是尽可能将最大数量的数据分配给模型参数估计。一种可能的策略是,使用特定的数据子集来确定哪些预测值是准确的。如果数据集不是特别大,那么在如何使用分配数据方面可能会有一些重叠,因此可靠的数据使用方式是重要的。
本节演示了基于不同目的的数据集的拆分。
2.1 拆分数据集的常用方法
经验模型验证的主要方法是将现有的数据集分为训练集和测试集。一部分数据被用来开发和优化模型,这些包含了大部分数据的是训练集。另一部分数据作为测试集,直到一两个模型被选出作为最可能的模型,测试集被用来作为最终仲裁器。
拆分数据集的方法往往是视具体情况而定的。假设将80%数据分给训练集,20%用于测试。常见的方法是简单随机抽样。rsample::initial_split()是为此创建的。它将数据框作为参数,将其放入训练中的比例。
# 设置随机数种子,以便日后可以重复结果
set.seed(501)
ames_split <- initial_split(ames, prop = 0.80)
ames_split
#> <Training/Testing/Total>
#> <2344/586/2930>
然而,ames_split只包含了分区的信息,为了获得结果的数据集,我们使用了另外的两个函数:
ames_train <- training(ames_split)
ames_test <- testing(ames_split)
dim(ames_train)
#> [1] 2344 74
许多情况下,简单随机抽样是合适的,但是也存在例外。当分类问题存在明显的类别不平衡时,一个类别的发生频率要明显高于或低于另一个分类的时候,使用简单随机抽样可能会将这些不常见的样本不成比例的分配到训练集或测试集中。为了避免此情况的发生,我们可以采用分层抽样,训练集和测试集在每个分类中进行抽取,然后进行整合。回归问题中,结果数据可以被人为的分为四分位数,然后进行四次不同的分层抽样。这样可以保证在训练集和测试集之间的分布相似。Ames数据的销售价格分布如图:
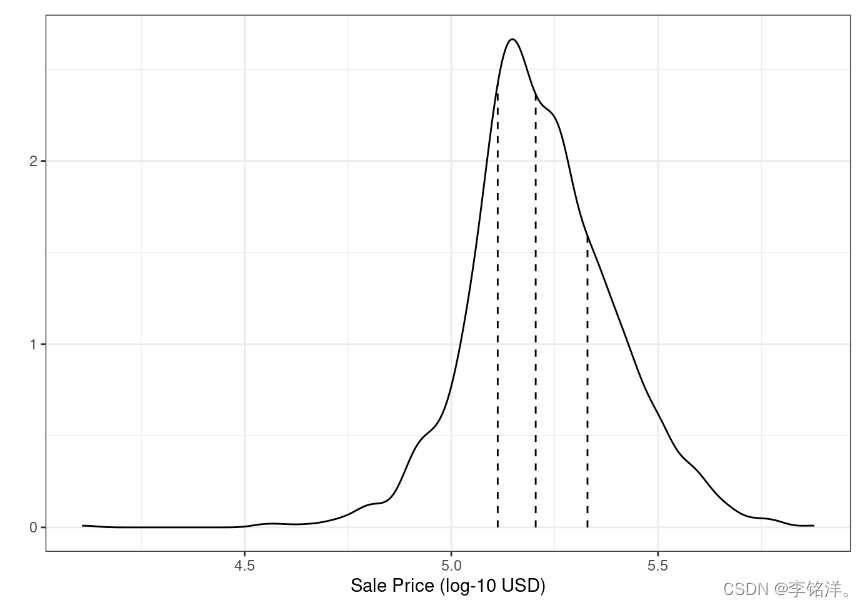
如前面讨论的,该销售价格是向右倾斜的。在此进行简单分割可能产生的担忧是,较为昂贵的房屋在训练集中不会得到很好的代表,这可能会增加模型在预测此类房产价格时的无效的风险。上图虚线把这些数据分为四个四分位数,分层随机样本在每个数据子集中进行80/20的分割,然后汇总结果。rsample中是使用strata参数实现的:
set.seed(502)
ames_split <- initial_split(ames, prop = 0.80, strata = Sale_Price)
ames_train <- training(ames_split)
ames_test <- testing(ames_split)
dim(ames_train)
#> [1] 2342 74
是否存在随机抽样不是最佳选择的情况呢?有的,一种情况是数据具有重要的时间成分,例如时间序列数据。这里,更常见的是使用最新的数据作为测试集。rsample包含一个initial_time_split()的函数,该函数与initial_split()类似。与随机抽样不同,属性参数prop表示将第一部分数据的多大比例用于训练集,该函数假定数据已经按照适当的顺序进行了预排序。
拆分数据的比例取决于具体问题。训练集数据太少会阻碍模型找到合适的参数估计的能力。相反测试集数据太多会降低性能估计的质量。统计界的一些人一般会避开测试集,因为他们认为所有数据都应该用于参数估计。虽然这一论点有可取之处,但将一组不偏不倚的观察结果作为模型质量的最终仲裁者是很好的建模实践。只有在数据非常小的情况下,才应该避免使用测试集。
2.2 验证集
描述拆分数据目标时,我们挑选测试集用于正确评估最终模型的性能,那么,如果在测试集之前想要测量性能,要怎么办?
经常听到的答案时使用验证集,特别是在神经网络和深度学习的文献中。在神经网络的早期,研究者就意识到通过重新预测训练集样本来衡量性能会导致过于乐观的结果(significantly,不切实际)。这导致了模型的过度拟合,这意味着它们在训练集表现很好但在测试集表现不佳。为了解决这个问题,一个小的验证数据集被保留下来,用于网络训练时测量性能。一旦验证集错误率开始上升,训练将停止。换句话说,验证集是一种在使用测试集之前粗略了解模型表现的方法。
验证集取自训练集的重采样的情况将在后续内容讨论。如果使用验证集,可以从不同拆分功能开始:
set.seed(52)
# 将60%作为训练,20%作为验证,20%作为测试:
ames_val_split <- initial_validation_split(ames, prop = c(0.6, 0.2))
ames_val_split
#> <Training/Validation/Testing/Total>
#> <1758/586/586/2930>
使用各数据集:
ames_train <- training(ames_val_split)
ames_test <- testing(ames_val_split)
ames_validation <- validation(ames_val_split)
2.3 多层次数据
从统计学上讲,一个属性的数据是独立于其他属性的,这样的多层次数据种进行数据拆分应该在数据的独立试验单位级别进行。
2.4 其他需考虑问题
首先,要将测试集与任何建模活动隔离开来。其次,对训练集进行二次采样可以缓解特定问题,比如可以让分类更加均衡,测试集会更加全面反映模型在测试集外遇到的情况。还有,不要对不同的任务使用相同的数据。
三、parsnip用于拟合模型
parsnip数据包是tidymodels元包中的R包之一。具体来讲,我们将重点介绍fit()和predict()。
3.1 创建模型
一旦数据被编码成为准备用于建模算法的格式,例如数字矩阵,它们就可以在模型构建过程中使用。
假设线性回归模型是我们最初的选择。这就指定了结果数据是numeric的且预测因素以简单的斜率和截距与结果相关:
y i = β 0 + β 1 + x 1 i + ⋯ + β p x p i y_{i}=\beta _{0}+\beta _{1}+x_{1i}+\dots+\beta _{p}x_{pi} yi=β0+β1+x1i+⋯+βpxpi
可以采用多种方法来估计模型参数:
-
普通线性回归采用传统的最小二乘法来求解模型参数
-
正则化线性回归在最小二乘法的基础上增加了惩罚项,以鼓励模型简化,减少预测变量或将它们的系数缩小至零。这可以通过贝叶斯或非贝叶斯的技术来实现。
补充说明:在机器学习和统计建模中,惩罚项是指在损失函数中添加的一项,用于对模型的复杂度进行惩罚或控制。它可以帮助防止过拟合(overfitting)或者在模型中选择重要的特征,促使模型更加简单和泛化能力更强。惩罚项通常是正则化方法(通过添加惩罚/控制项限制模型参数的大小和数量)的一部分,常见的有L1范数和L2范数,分别对应Lasso和Ridge回归中的惩罚项。L1正则化(Lasso):在损失函数中增加模型参数的L1范数(参数的绝对值之和)作为惩罚项。这种方法倾向于使得部分参数变为零,因此可以用于特征选择。L2正则化(Ridge):在损失函数中增加模型参数的L2范数(参数的平方和)作为惩罚项。这种方法倾向于让参数的值变小但不一定为零,有助于减少参数的大小。通过调整正则化项的权重,可以控制模型对复杂性的敏感程度。这些方法在线性回归、逻辑回归和其他机器学习模型中广泛应用,有助于提高模型的泛化能力并避免过度拟合训练数据。
R中,stats包可以用于情况1,使用lm()函数的线性回归语法是:
model <- lm(formula, data, ...)
为了使用正则化进行估计,情况可以使用rstanarm包进行拟合Bayesian模型:
model <- stan_glm(formula, data, family = "gaussian", ...)
他和lm()一样只有formula项可用。
有种流行的非贝叶斯正则化回归方法是glmnet模型:
model <- glmnet(x = matrix, y = vector, family = "gussian", ...)
注意,这些函数接口再数据传给模型函数或以其参数方面是异质的。问题一在于,需要拟合不同的包装的模型,必须以不同的方式对数据进行格式化。问题二在于,不同函数接口不同。对于试图进行数据分析的人,需要记住每个软件包的语法,这会非常令人沮丧。
对于tidymodels,指定模型的方法旨在更加统一:
-
根据其数学结构(如线性回归、随机森林、KNN等)指定模型的类型。
-
指定用于拟合模型的工程。大多数情况下,这反映的是应该使用的软件包,如
Stan或glmnet。这些模型本身就是模型,parsnip通过将他们用于建模引擎来提供一致的接口。 -
如果需要,要声明模型的模式。该模式反应了预测结果的类型。对于数字结果,模式是回归(regression);对于定性结果,模式是分类(classifiction)。如果模型算法只能解决一种类型的预测结果,例如线性回归,则已经设置了模式。
这些规范是在不参考数据的情况下构建的。例如,对于我们概述的三个案例:
linear_reg() |> set_engine("lm")
#> Linear Regression Model Specification (regression)
#>
#> Computational engine: lm
linear_reg() |> set_engine("glmnet")
#> Linear Regression Model Specification (regression)
#>
#> Computational engine: glmnet
linear_reg() |> set_engine("stan")
#> Linear Regression Model Specification (regression)
#>
#> Computational engine: stan
一旦制定了模型的详细信息,就可以使用fit()函数(使用formula)或者fit_xy()函数(当数据已经预处理)进行模型估计。parsnip包让用户不考虑底层接口,即使建模包只有x/y接口,也可以使用formula。
translate()函数可以展示出parsnip代码把用户代码转换为各种包的语法的详情:
linear_reg() |> set_engine("lm") |> translate()
#> Linear Regression Model Specification (regression)
#>
#> Computational engine: lm
#>
#> Model fit template:
#> stats::lm(formula = missing_arg(), data = missing_arg(), weights = missing_arg())
linear_reg(penalty = 1) |> set_engine("glmnet") |> translate()
#> Linear Regression Model Specification (regression)
#>
#> Main Arguments:
#> penalty = 1
#>
#> Computational engine: glmnet
#>
#> Model fit template:
#> glmnet::glmnet(x = missing_arg(), y = missing_arg(), weights = missing_arg(),
#> family = "gaussian")
linear_reg() |> set_engine("stan") |> translate()
#> Linear Regression Model Specification (regression)
#>
#> Computational engine: stan
#>
#> Model fit template:
#> rstanarm::stan_glm(formula = missing_arg(), data = missing_arg(),
#> weights = missing_arg(), family = stats::gaussian, refresh = 0)
说明:我们为glmnet提供了penalty参数。此外,对于stan和glmnet引擎,family参数被自动添加为默认参数,后续可以更改。
下面举个例子,让我们看看如何仅根据经纬度来预测Ames数据中的房屋销售价:
lm_model <-
linear_reg() |>
set_engine("lm")
lm_form_fit <-
lm_model |>
fit(Sale_Price ~ Longitude + Latitude, data = ames_train)
lm_xy_fit <-
lm_model |>
fit_xy(
x = ames_train |> select(Longitude, Latitude),
y = ames_train |> pull(Sale_Price)
)
lm_form_fit
#> parsnip model object
#>
#>
#> Call:
#> stats::lm(formula = Sale_Price ~ Longitude + Latitude, data = data)
#>
#> Coefficients:
#> (Intercept) Longitude Latitude
#> -313.623 -2.074 2.965
lm_xy_fit
#> parsnip model object
#>
#>
#> Call:
#> stats::lm(formula = ..y ~ ., data = data)
#>
#> Coefficients:
#> (Intercept) Longitude Latitude
#> -313.623 -2.074 2.965
parsnip不仅为不同的包提供了一致的模型接口,而且提供了模型参数的一致性。适合同一模型的不同函数具有不同参数名称是很常见的。随机森林就是一个很好的例子。三个常用参数是集合中树的数量、树内每个分裂时的随机采样的预测值的数目以及进行分裂所需的数据点的数目。对于不同的R包,参数如下表所示:
| 参数类型 | ranger | randomForest | sparklyr |
|---|---|---|---|
| #预测值采样 | mtry | mtry | feature_subset_stratey |
| #树 | num.trees | ntree | num_trees |
| #分裂数据点 | min.node.size | nodesize | min_instances_per_node |
为减少参数指定的麻烦,parsnip在包内与包之间使用通用的参数,下表展示了随机森林中的参数使用:
| 参数类型 | parsnip |
|---|---|
| #预测值采样 | mtry |
| #树 | trees |
| #分裂数据点 | min_n |
parsnip将一些原始参数名进行了更改。比如有些原始参数名称可能会很专业,比如为了指定glmnet模型中的正则化量,使用了希腊字母lambda,对于许多人来说这个lambda代表了什么并不明显,由于其代表的是正则化中使用的惩罚(penalty)所以parsnip将其重命名为了penalty。类似地,KNN模型中的邻居的数量称为k而parsnip将其改回了neighbors。其大致命名规则就是如此人性化。
为了理解parsnip参数名称是如何映射到原始名称的,可以使用模型帮助文件(如?rand_forest)以及使用translate()函数:
rand_forest(trees = 1000, min_n = 5) |>
set_engine("ranger") |>
set_mode("regression") |>
translate()
#> Random Forest Model Specification (regression)
#>
#> Main Arguments:
#> trees = 1000
#> min_n = 5
#>
#> Computational engine: ranger
#>
#> Model fit template:
#> ranger::ranger(x = missing_arg(), y = missing_arg(), weights = missing_arg(),
#> num.trees = 1000, min.node.size = min_rows(~5, x), num.threads = 1,
#> verbose = FALSE, seed = sample.int(10^5, 1))
parsnip中建模函数将模型参数分为了两类:
-
主参数比较常用,往往可以跨引擎使用。
-
引擎参数要么用于特定的引擎,要么很少使用。
比如前面的随机森林代码的转换中,默认情况添加了参数num.threads,verbose,seed。这些参数特定于随机森林模型中,并不是主要参数。可以在set_engine()中指定特定于引擎的参数。例如,要让ranger::ranger()函数打印出有关配合的详细信息:
rand_forest(trees = 1000, min_n = 5) |>
set_engine("ranger", verbose = TRUE) |>
set_mode("regression")
#> Random Forest Model Specification (regression)
#>
#> Main Arguments:
#> trees = 1000
#> min_n = 5
#>
#> Engine-Specific Arguments:
#> verbose = TRUE
#>
#> Computational engine: ranger
3.2 使用模型结果
创建拟合了模型后,我们就可以从各种方式使用结果;我们可能想要绘制、打印或以其他方式检查模型输出。几个量存储在parsnip模型对象中,包括拟合的模型。这可以在一个名为fit的元素中找到,它可以使用extract_fit_engine()函数返回:
lm_form_fit |> extract_fit_engine()
#>
#> Call:
#> stats::lm(formula = Sale_Price ~ Longitude + Latitude, data = data)
#>
#> Coefficients:
#> (Intercept) Longitude Latitude
#> -313.623 -2.074 2.965
可以对此对象应用常规方法如打印或绘图:
lm_form_fit |> extract_fit_engine() |> vcov()
#> (Intercept) Longitude Latitude
#> (Intercept) 273.852441 2.052444651 -1.942540743
#> Longitude 2.052445 0.021122353 -0.001771692
#> Latitude -1.942541 -0.001771692 0.042265807
Base R中一些现有方法的一个问题是,结果的存储方法可能不是最有用的。比如,lm对象的summary()方法可以用于打印模型拟合的结果,其中包含参数值、它们的不确定性估计和p值。还可以保存这些特定结果:
model_res <-
lm_form_fit |>
extract_fit_engine() |>
summary()
# 模型系数表可以通过`coef`方法访问
param_est <- coef(model_res)
class(param_est)
#> [1] "matrix" "array"
param_est
#> Estimate Std. Error t value Pr(>|t|)
#> (Intercept) -313.622655 16.5484876 -18.95174 5.089063e-73
#> Longitude -2.073783 0.1453353 -14.26896 8.697331e-44
#> Latitude 2.965370 0.2055865 14.42395 1.177304e-44
这一结果中,需要注意几点。首先,对象是一个数字矩阵。选择该数据结构很可能是因为所有的计算结果都是数值的,并且矩阵对象的存储效率比数据框更高。这一选择可能是在20世纪70年代末做出的,当时的计算效率非常的关键。其次,非数字数据包含在了行名中,这种行为非常复合原始S语言中的约定。
合理的下一步操作可能是创建参数值的可视化。要做到这一点,将参数矩阵转换为数据框是需要的。我们可以将行名作为列添加,以便可以在绘图中使用它们。然而,需要注意的是,一些现有的矩阵列名不是普通数据框的有效R列名(如"Pr(>|t|)")。另一个复杂的问题是列名的一致性。比如对于lm对象来说,p值的列名是"Pr(>|t|)"但是对于其他模型,可能就不是了(“Pr(>|z|)”)。
作为一种解决方案,broom包可以将许多类型的模型对象转换为整洁的结构。例如,使用tidy():
tidy(lm_form_fit)
#> # A tibble: 3 × 5
#> term estimate std.error statistic p.value
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 (Intercept) -314. 16.5 -19.0 5.09e-73
#> 2 Longitude -2.07 0.145 -14.3 8.70e-44
#> 3 Latitude 2.97 0.206 14.4 1.18e-44
3.3 做出预测
另一个parsnip与传统R建模函数不同的方面是predict()返回值的格式。对于预测来说,parsnip始终遵循以下规则:
-
结果是一个tibble
-
tibble的列名是可预测的
-
tibble的行数始终与输入数据集中的行数相同
比如:
ames_test_samll <- ames_test |> slice(1:5)
predict(lm_form_fit, new_data = ames_test_samll)
#> # A tibble: 5 × 1
#> .pred
#> <dbl>
#> 1 5.23
#> 2 5.29
#> 3 5.28
#> 4 5.27
#> 5 5.27
预测的行序始终与原始数据相同。
这三条规则使预测与原始数据的合并变得更加容易:
ames_test_samll |>
select(Sale_Price) |>
bind_cols(predict(lm_form_fit, ames_test_samll)) |>
bind_cols(predict(lm_form_fit, ames_test_samll, type = "pred_int"))
#> # A tibble: 5 × 4
#> Sale_Price .pred .pred_lower .pred_upper
#> <dbl> <dbl> <dbl> <dbl>
#> 1 5.33 5.23 4.92 5.54
#> 2 5.29 5.29 4.98 5.60
#> 3 5.25 5.28 4.97 5.59
#> 4 5.27 5.27 4.97 5.58
#> 5 5.33 5.27 4.96 5.58
规则2下的tidymodels,不同类型的预测的可预测名如下表所示:
| type value | column names |
|---|---|
| numeric | .pred |
| class | .pred_class |
| prob | .pred_{class levels} |
| conf_int | .pred_lower, .pred_upper |
| pred_int | .pred_lower, .pred_upper |
这些规则使得数据分析过程和语法同质化,使用户可以把时间花费在结果上,不必关注R包之间的语法差异。
3.4 parsnip扩展包
parsnip包本身包含许多模型的接口。然而,为了便于软件包的安装与维护,还有其他的tidymodels包,为其他模型提供了parsnip包的模型定义。discrim包有一组分类技术的模型定义,称为判别分析方法。通过这种方式减少了安装parsnip包所需要的包依赖性。可以在http://www.tidymodels.org/find上查找可以与parsnip一起使用的模型的列表。
3.5 创建模型的型号与规则快捷
记住许多模型的编写规则可能比较枯燥乏味。parsnip提供了一个RStudio addin帮助:
parsnip_addin()
这将会在RStudio IDE查看面板打开一个窗口,其中包含了每个模型模式的可能列表,可以直接写入源代码面板。
总结
至此,我们学习了tidymodels包中进行数学建模的基础做法。包括了如何对数据集进行拆分以用于模型拟合,数据可以根据需要拆分为训练集、测试集和验证集。在数据拆分的时候可以根据数据结构的不同,对数据进行不同层次的划分。当进行数学建模时,通过parsnip包可以使得建模过程更加的同质化,这就是tidymodels的优势,它可以让用户更加关注建模的结果,而不是把心思浪费在学习如何使用建模函数的过程上。










