1、前言
3D图像分割一直是医疗领域的难题,在这方面nnunet已经成为了标杆,不过nnunet教程较少,本人之前跑了好久,一直目录报错、格式报错,反正哪里都是报错等等。并且,nnunet对于硬件的要求很高,一般的电脑配置或者低配置的服务器完全带不起来
或者定义conv.3D的unet网络模型,但对显卡的要求也很高...
之前实现了unet的自适应多类别分割任务,博文如下
Unet 实战分割项目、多尺度训练、多类别分割_unet进行多类分割-CSDN博客
代码根据数据集的mask,可以自动计算出mask前景的类别,这样就能为unet的输出自动调整,不需要更改别的操作。
而3d的图像其实就是2d拼接起来的,或许可以将nii格式的3d图片切分,这样根据上文的代码就可以实现医疗图像3d的分割
2、 nii 文件的切分
import SimpleITK as sitk
这里用itk 对3d数据进行读取
2.1 数据集
这里的3d数据是 BRATS 脑肿瘤分割数据(brain tumor segmentation challenge,BraTS Chanllenge),这里只对训练集进行操作
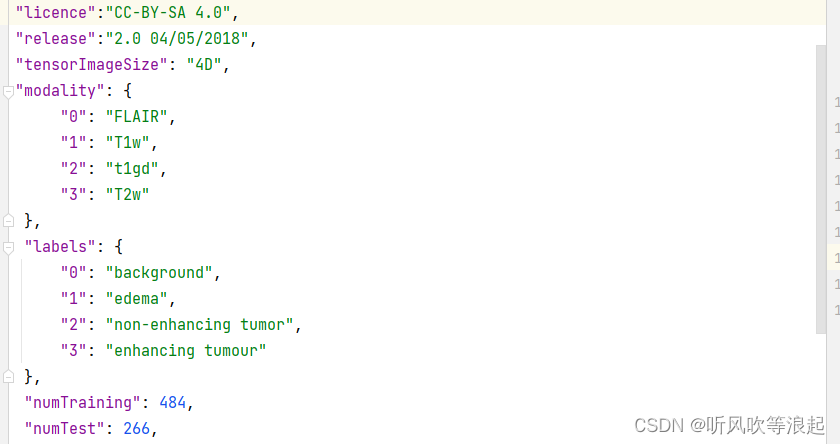
需要注意的是,一般的nii图像都是3D的,这里数据是4D的,好像是每个3D图像的模态,类似于官方的增强?
T1 成像,利于观察解剖结构,病灶显示不够清晰
T1gd 在受试者做磁共振之前向血液内注射造影剂,使成像中血流活跃的区域更加明显,是增强肿瘤的重要判据
T2 成像,病灶显示较为清晰,判断整颗肿瘤
FLAIR(抑制脑脊液的高信号),含水量大则更亮眼,可以判断瘤周水肿区域

mask模板是四分类的:

2.2 slice 切片代码
代码放在这里:
这里参考之前的博文:nii 文件的相关操作(SimpleITK)_如何使用nii文件做深度学习-CSDN博客
import SimpleITK as sitk
import numpy as np
import os
from tqdm import tqdm
import shutil
import cv2
# 新建目录
def mkdir(rt):
ret_path = rt + '_ret2D'
if os.path.exists(ret_path): # 删除之前的切片目录
shutil.rmtree(ret_path)
os.mkdir(ret_path)
os.mkdir(os.path.join(ret_path,'images'))
os.mkdir(os.path.join(ret_path,'labels'))
def get_image_from_nii(x,y,name,thre): # 传入nii文件,对nii进行切片
img = sitk.ReadImage(x)
img_array = sitk.GetArrayFromImage(img) # nii-->array
label = sitk.ReadImage(y)
label_array = sitk.GetArrayFromImage(label) # nii-->array
for index,i in enumerate(range(img_array.shape[1])): # TODO 需要根据img维度更改,4D设定为1,3D设置为0
img_select = img_array[0,i, :, :] # TODO 需要根据img维度更改,从x轴切分,[:,i,:]从y轴切分
label_select = label_array[i, :, :]
# 图片保存目录
img_save_name = os.path.join(root+'_ret2D','images',name+'_'+str(index)+'.png')
label_save_name = os.path.join(root+'_ret2D','labels',name+'_'+str(index)+'.png')
h,w = label_select.shape
total_pixel = h*w # 总的像素点个数
if label_select.max() == 0: # 没有前景的像素点不保存
continue
else:
# 归一化
img_select = (img_select - img_select.min()) / (img_select.max() - img_select.min())*255
img_select = img_select.astype(np.uint8)
label_select = label_select.astype(np.uint8)
if (np.sum(label_select !=0 ) / total_pixel) > thre:
cv2.imwrite(img_save_name,img_select)
cv2.imwrite(label_save_name,label_select)
# 切片函数
def sliceMain(rt,imgf,labf,thre):
# 删除之前的切片目录,建立新的目录
mkdir(rt)
nii_list = [i for i in os.listdir(os.path.join(rt,imgf))]
for image_nii in tqdm(nii_list): # 遍历所有的nii文件
name = image_nii.split('.nii.gz')[0]
image_nii = os.path.join(rt,imgf,image_nii)
label_nii = image_nii.replace(imgf,labf) # 自动获取nii 的标签
get_image_from_nii(image_nii,label_nii,name,thre)
if __name__ == '__main__':
root = 'BRATS' # 待切分nii文件的父目录
images_folder = 'imagesTr' # 3d nii的数据
labels_folder = 'labelsTr' # 3d nii 的标签数据
threshold = 0.03 # 分割的比例不超过阈值的数据删除
# 切片函数
sliceMain(
rt=root,
imgf=images_folder,
labf=labels_folder,
thre=threshold
)
这里简单介绍一下:目录结构如下

具体数据的名称和后缀要严格对应!!!


threshold 是阈值处理,如果mask前景的像素点个数没有达到整个图片像素点的阈值,就不会被保存。这里默认是0.03

切分的时候,因为这里是4D的,所以img_array是四维的,我们默认取第一个维度的3D图像
同时,3D图像可以用x,y,z三个坐标表示,这里的shape1就是沿着x轴进行2D的切分

因为医学图像的灰度动态范围很多,可能到上千,因此这里将灰度值重新映射,变成np的uint8格式,再用cv保存

2.3 保存格式
图像的保存,这里搞了好久,要么格式问题,要么灰度有问题。这里做下总结
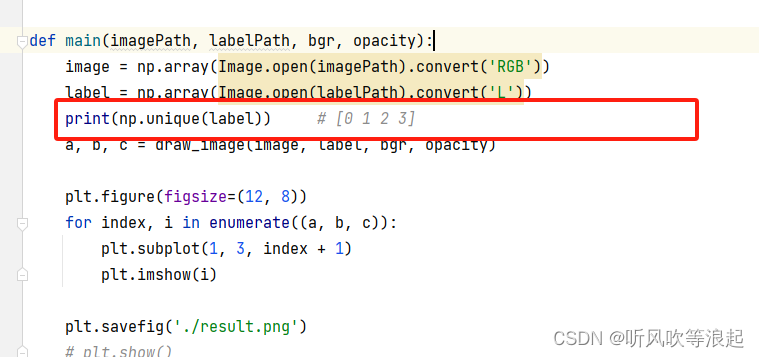
首先,png格式可以完整的保存2D切分的信息,而不会因为图像压缩导致mask灰度值改变。说人话就是,这里切分的2d像素值只有0 100 255,如果保存为其他格式,可能读取的时候,会产生0 1 2 3....等等灰度图像,而分割的mask是阈值图像!!
其次,plt保存的时候,会将图像重新映射,我们只想要0 1 2这种格式,但是他可能会把0变成0,1变成128.2变成255这样。虽说,这样看mask确实方便,不至于变成全黑的,但是本人测试的时候,总会莫名多出一个灰度。说人话就是,本来这里是四分类的,plt保存的时候,np.unique读取的时候,会变成5个类别
这里搞了半天,本人电脑太差,测试半天,只有这个代码是符合的。至于问题到底是不是我说的那样,可以自己测试
代码如下:
import os
from tqdm import tqdm
import numpy as np
import cv2
root = './BRATS_ret2D/labels' # 训练 mask的路径
masks_path = [os.path.join(root ,i) for i in os.listdir(root)]
gray = [] # 前景像素点
for i in tqdm(masks_path,desc="gray compute"):
img = cv2.imread(i,0)
img_uni = np.unique(img) # 获取mask的灰度值
for j in img_uni:
if j not in gray:
gray.append(j)
print(gray)

2.4 切分好的数据
上述代码,切分后会生成root的返回目录

这里的mask并不是全黑的,只是0 1 2 3这样导致很黑而已。这里的目录名称按照切分索引,而没有从0开始,这样就能看出来BRATS_001 里面,49之前的要么没有mask前景,要么前景的区域不足我们设定的阈值!


3、划分数据集
参考之前的代码:关于图像分割任务中按照比例将数据集随机划分成训练集和测试集_图像分割数据集怎么划分-CSDN博客

这里可以可视化一下:关于图像分割项目的可视化脚本-CSDN博客

4、训练
unet训练如下:
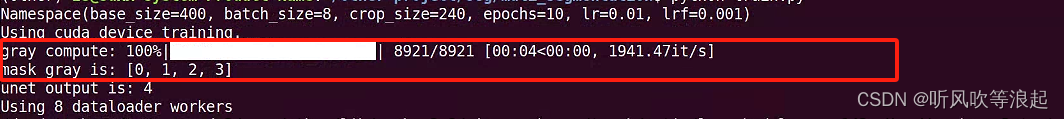
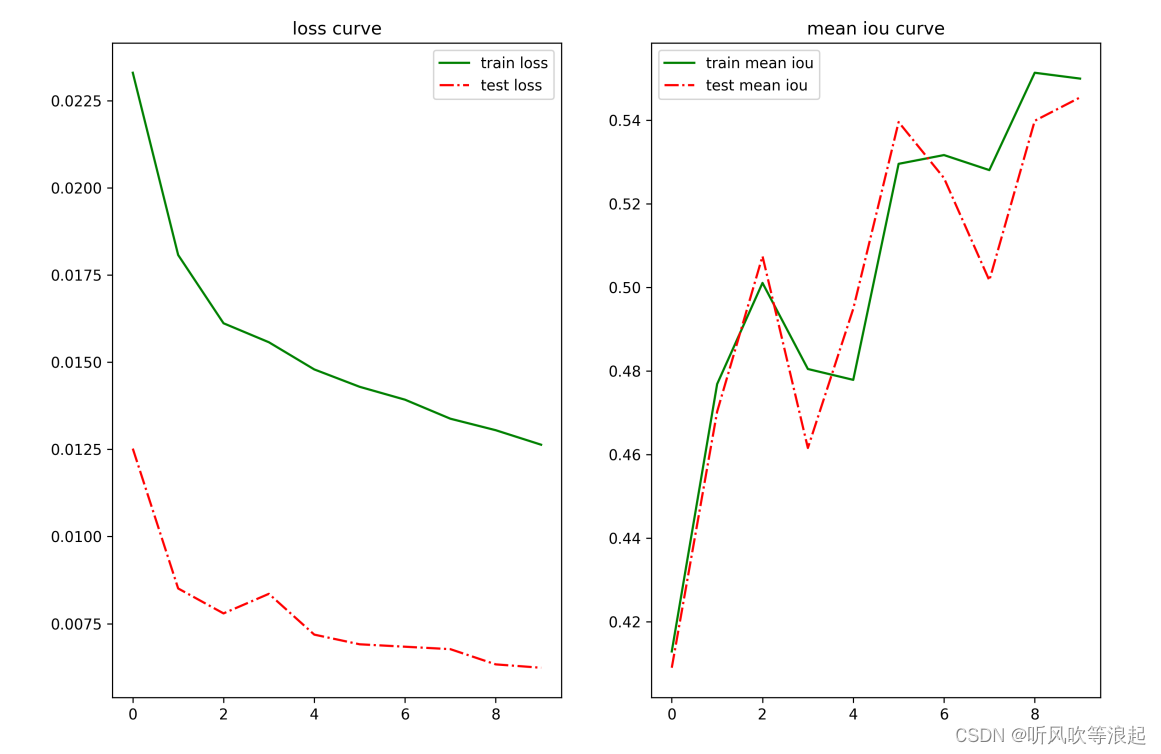
训练时间太长了, 这里只简单训练了10个epoch用作测试,结果如下:
代码是这篇的代码:Unet 实战分割项目、多尺度训练、多类别分割_unet进行多类分割-CSDN博客
训练日志里面,有每个类别的指标:

推理结果:

4、项目总结
1、准备好3D的nii.gz数据,然后根据本章第二节摆放好数据切分。根据项目的实际要求设定好阈值或者沿着哪个轴切分
2、划分数据很简单
3、训练的 train 脚本
4、推理的时候,把待推理的数据放在inference目录下即可
5、说点废话
对于项目的改进的思考,项目下载:
深度学习Unet实战分割项目:BraTS3d脑肿瘤图像切分的2D图片分割项目(4分类)资源-CSDN文库
因为医学图像的灰度值都很低,往往图像会很暗,这样图像的梯度信息啊、边缘信息啊都很模糊,效果不太好,可以利用医学图像常用的windowing方法,其实就是对比度拉伸
医学图像处理的windowing 方法_医学图像常用windowing和histogram equalization-CSDN博客
而且,不同于正常的分类图像,这里的normalize可能直接 - 0.5 在除以 2效果不太好,这可以手动计算好图像的mean和std,可以有效提升网络的性能
怎么计算数据的均值和方差_计算数据集均值和方差-CSDN博客










