目录
一、概述
二、算例及仿真
📢算例一:
📢算例二:
📢算例三:
📢算例四:
📢算例五:
📢算例六:
三、Matlab代码实现
一、概述
本文通过结合线性时不变滤波器、正交多分辨率表示和基于稀疏性的方法,解决了处理批处理模式时间序列数据时的信号去噪和模式识别问题。利用数字滤波器状态空间表示的频谱变换,将高阶零相低通、高通和带通无限脉冲响应滤波器设计为矩阵的新方法。还提出了一种基于近端梯度的技术,用于对一类特殊的零相位高通和带通数字滤波器进行因式分解,以便因式分解积保持滤波器的零相性质,并在信号模型中加入输入的稀疏导数分量。为了展示本文新颖的滤波器设计的应用,验证并提出了新的信号模型,以同时去噪和识别感兴趣的模式。首先使用我们提出的滤波器设计来测试现有的信号模型,该模型同时结合了线性时间不变(LTI)滤波器和基于稀疏性的方法。将本文提出的滤波器设计与现有信号模型相结合,开发了一种称为稀疏性辅助信号去噪(SASD)的新信号模型。使用仿真数据,证明了SASD信号模型在不同阶次滤波器和噪声水平下的鲁棒性。此后,提出并推导出一种称为稀疏性辅助模式识别(SAPR)的新信号模型。在SAPR中,还将LTI带通滤波器和基于稀疏性的方法与正交多分辨率表示(如小波)相结合,以检测输入信号中的特定模式。最后,本文将信号去噪和模式识别任务相结合,并推导出一种称为稀疏性辅助信号去噪和模式识别(SASDPR)的新信号模型。分别使用睡眠脑电图数据来检测K复合物和睡眠纺锤体,从而说明了SAPR和SASDPR框架的功能。
二、算例及仿真
📢算例一:
稀疏辅助信号去噪( SASD )算法同时结合全变差去噪和低通滤波对含噪信号进行滤波,从而保持了信号的不连续性。
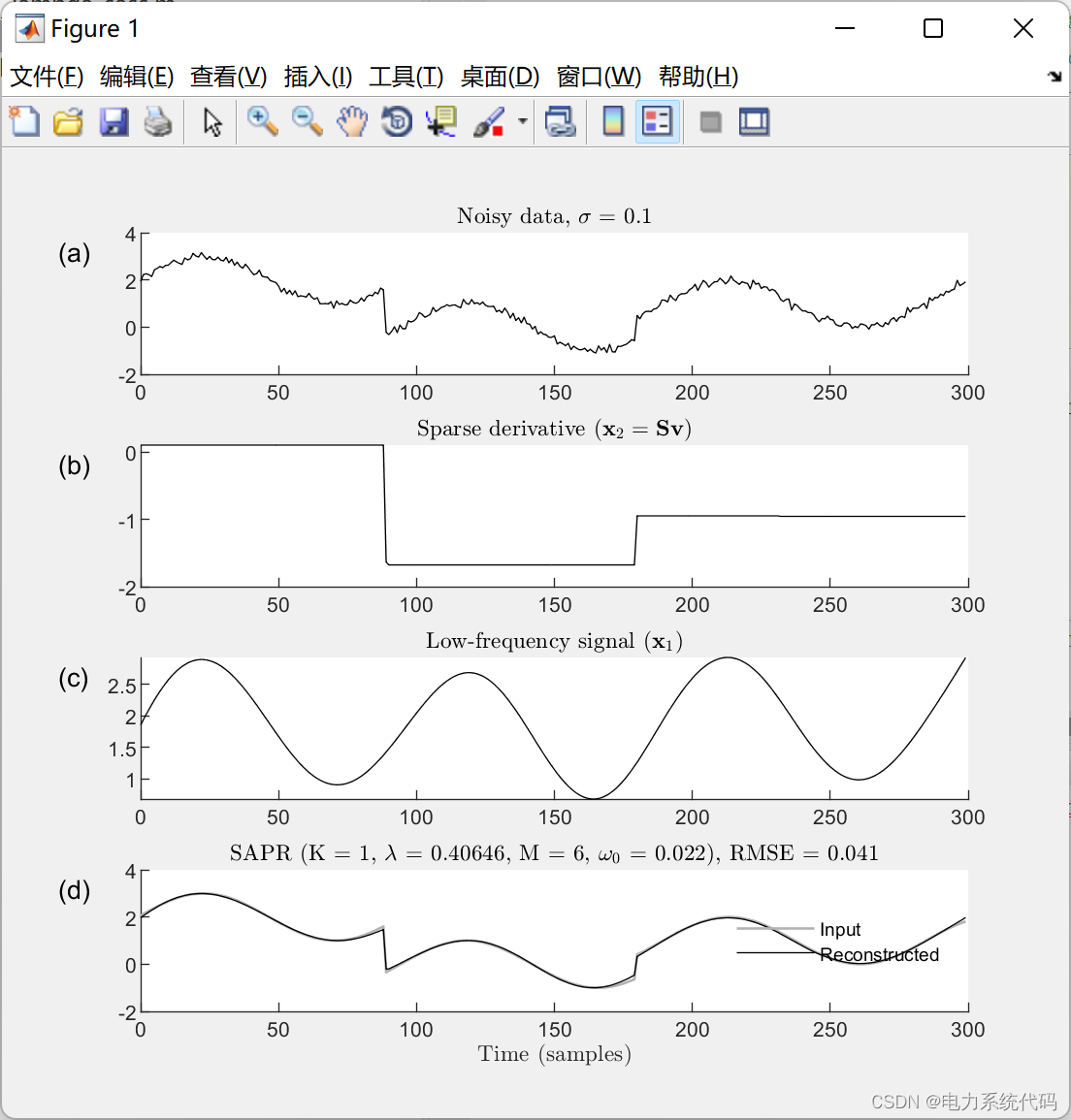
📢算例二:
使用带状矩阵演示零相位滤波的示例:
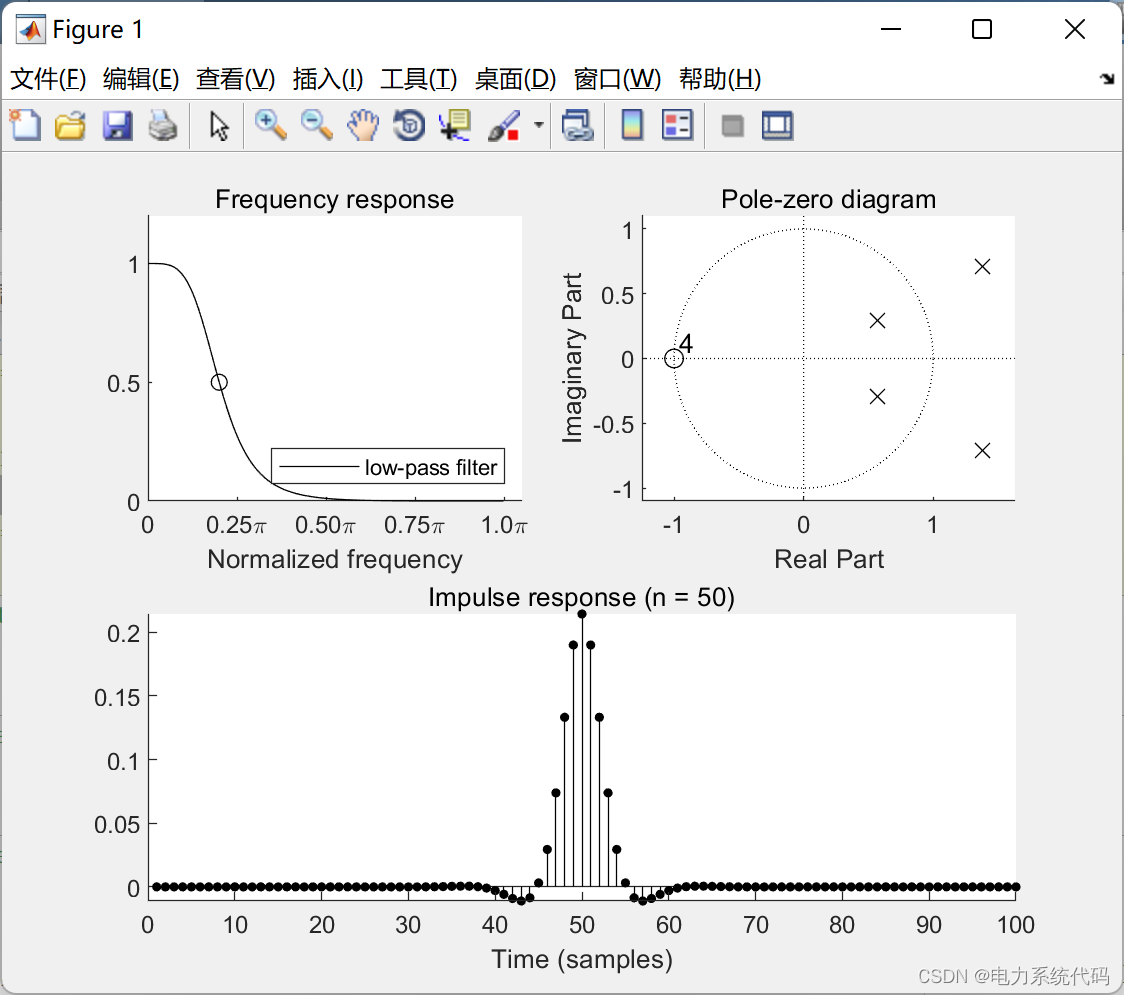
📢算例三:
稀疏辅助信号去噪( SASD )算法同时结合全变差去噪和低通滤波对含噪信号进行滤波,从而保持了信号的不连续性。在这个例子中,我们使用LPF、TVD、SASS和SASD对心电信号进行去噪:
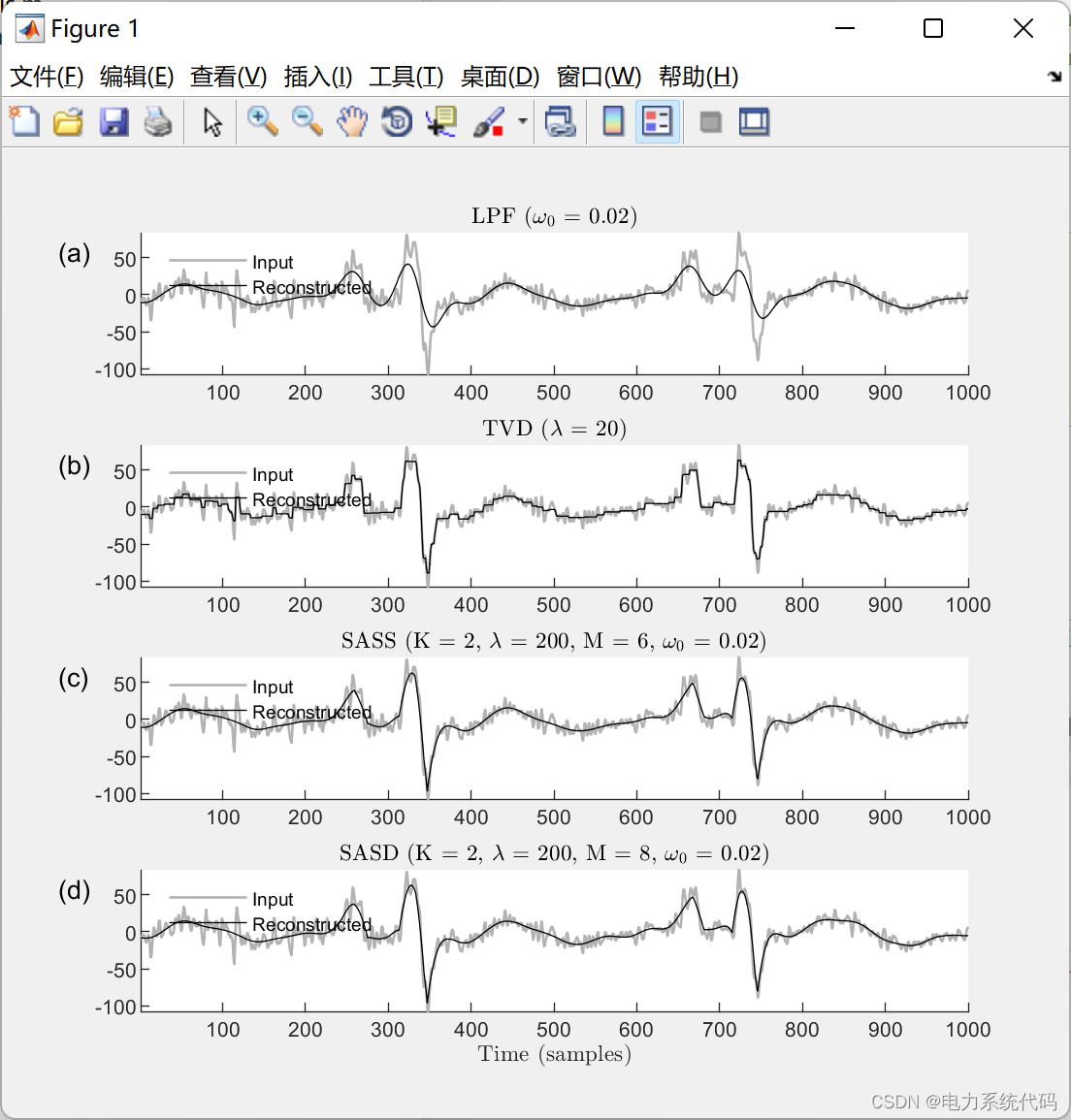
📢算例四:
稀疏辅助信号去噪和模式识别 (SASDPR) 将同时去噪和检测给定信号中感兴趣的振荡模式:
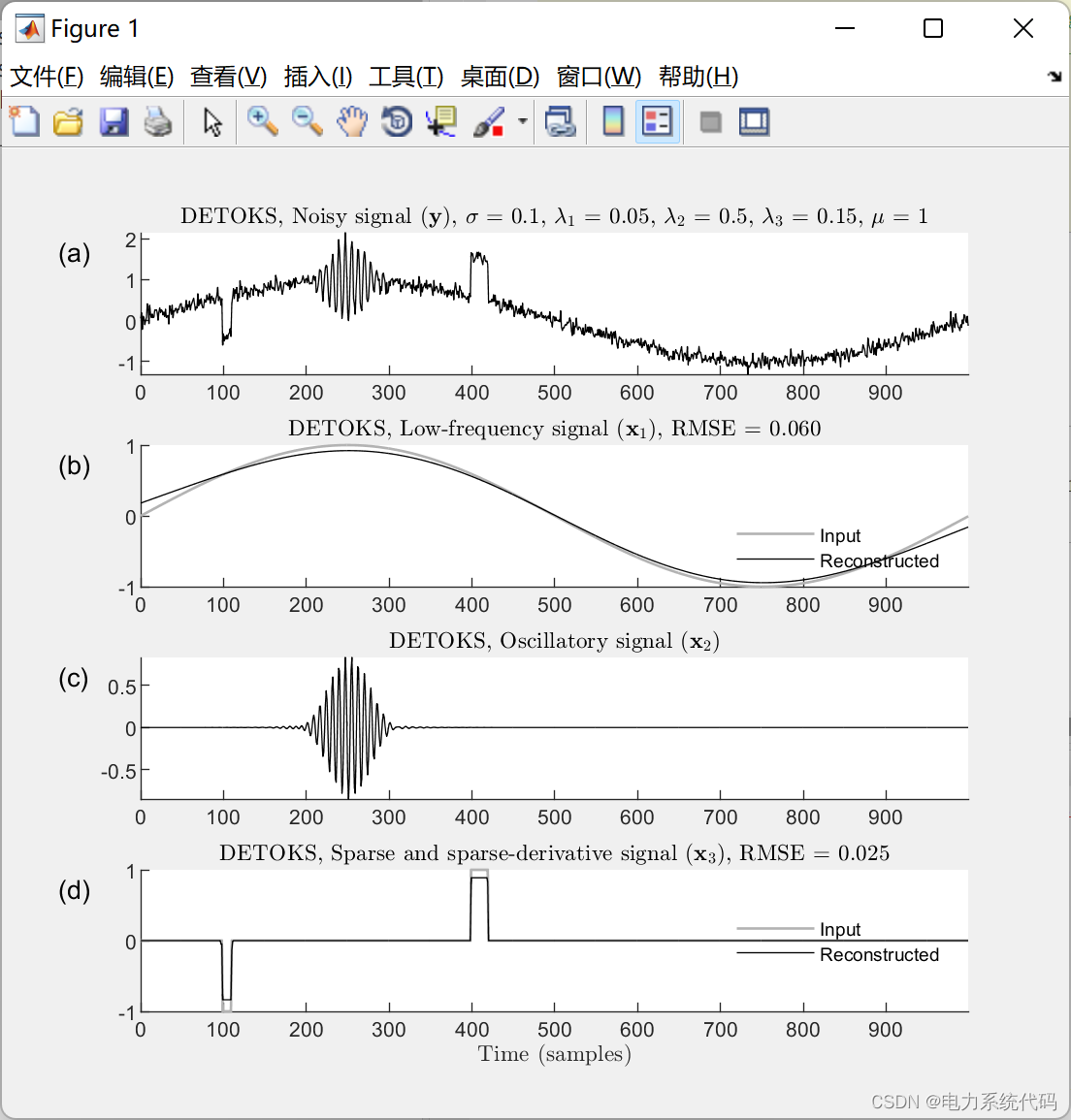
📢算例五:
稀疏辅助信号去噪和模式识别 (SASDPR) 将同时去噪和检测给定信号中感兴趣的振荡模式:
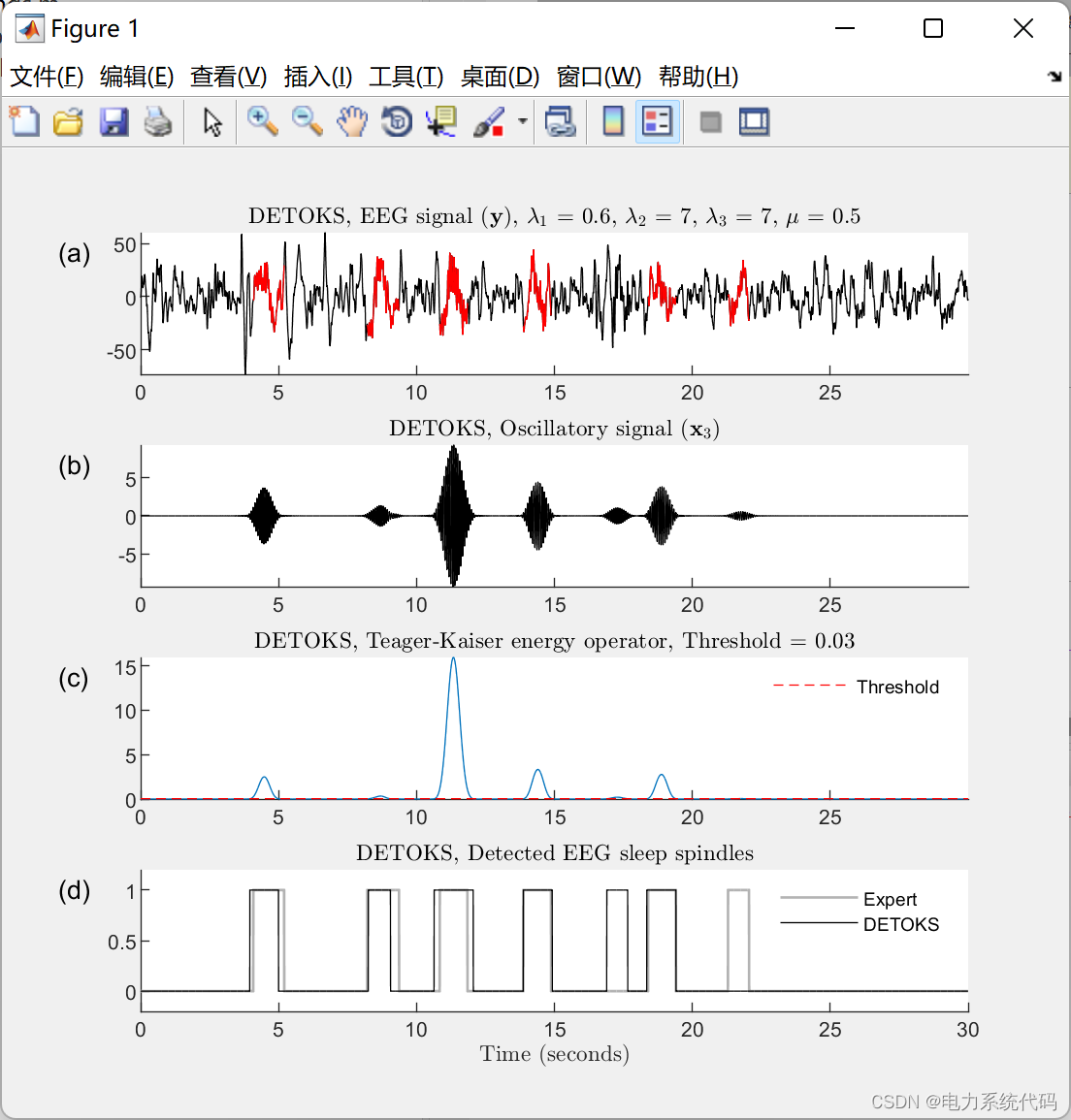
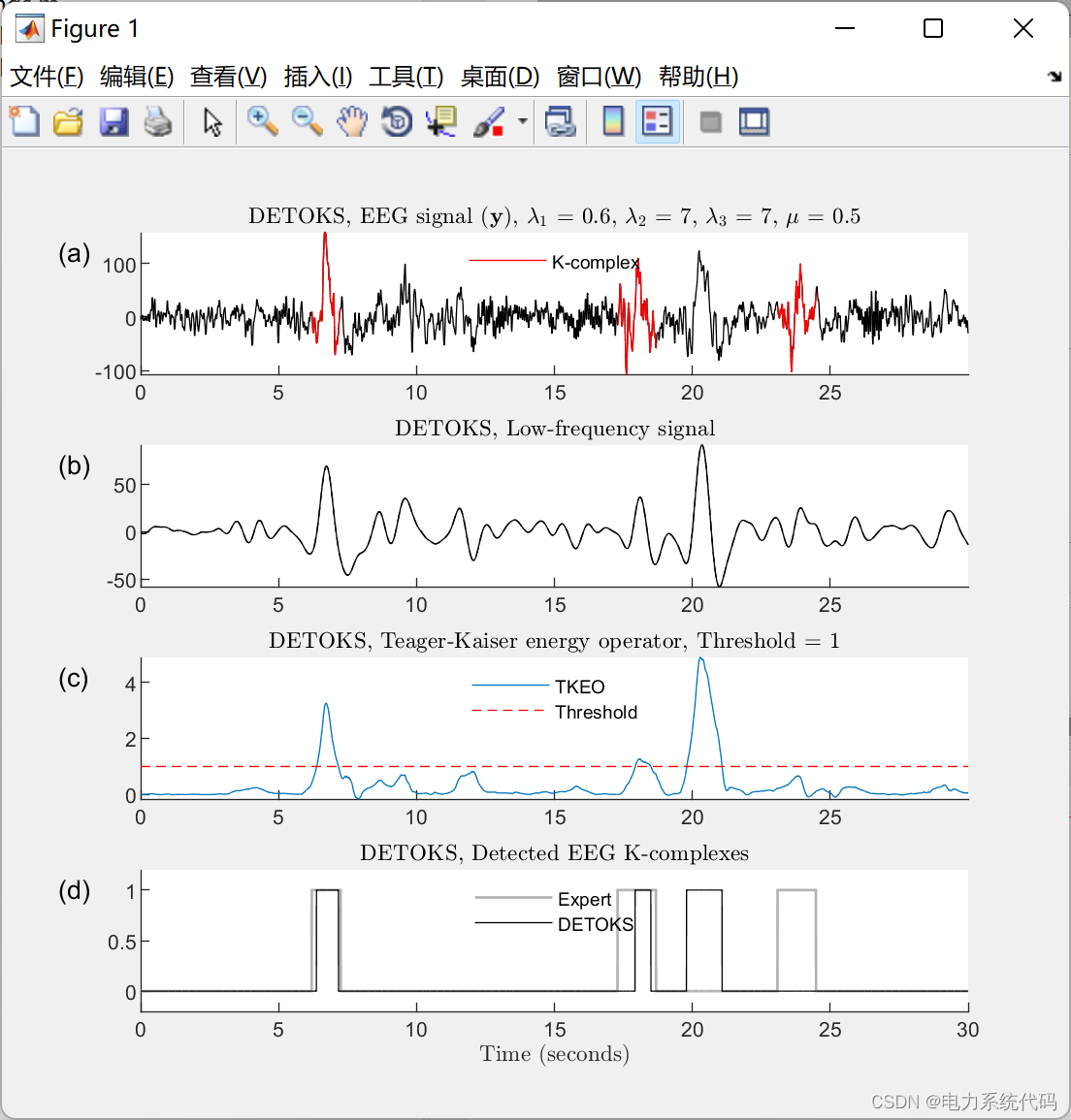
📢算例六:
稀疏辅助模式识别 (SAPR) 将检测信号中的小波模式:
三、Matlab代码实现
本文仅展现部分代码,全部代码点这里:
function [y, f, s, x, w] = generate_signal( fs, sigma )
%% 初始化噪声电平和信号长度
rng('default')
N = 10*fs;
n = 0:N-1;
%% 产生低频合成成分
f = 0.1;
f = sin(2*f/fs*pi*n);
%% 振荡
s = zeros(size(n));
o = 13;
s(200+(1:fs)) = sin(2*pi*o/fs*(1:fs)) .* hamming(fs)';
%% 稀疏分段常数
x = zeros(size(n));
x(100:110) = -1;
x(400:420) = 1;
%% 添加噪音
w = sigma*randn(size(n));
y = f+s+x+w;
end
%% plot_signals( y, x_ex3, kc, k, tk, th, binX, binA, method)
%
% Inputs:
% y - 噪声信号
% x_ex3 - 睡眠纺锤信号
% k - 检测到k-综合波信号
% tk - Teager-Kaiser能源运营商
% th - 主轴检测阈值
% binX - 二进制向量
% binA - 带有算法注释主轴的二进制向量
% method - SASDPR/DETOKS
%
% Outputs:
% None
%% ________________________________________________________________________
%%
function plot_signals( y, x_ex3, kc, k, tk, th, binX, binA, method)
global simdata;
fs = simdata.fs;
if strcmp(method, 'DETOKS')
txt_1 = [method, ', EEG signal ($\mathbf{y}$), ','$\lambda_1$ = ', num2str(simdata.lam0_detoks), ...
', $\lambda_2$ = ', num2str(simdata.lam1_detoks), ', $\lambda_3$ = ', num2str(simdata.lam2_detoks),...
', $\mu$ = ', num2str(simdata.mu_detoks)];
txt_2 = [method, ', Low-frequency signal'];
txt_3 = [method, ', Teager-Kaiser energy operator, Threshold = ', num2str(simdata.th_detoks)];
txt_4 = [method, ', Detected EEG K-complexes'];
elseif strcmp(method, 'SAPR')
txt_1 = [method, ', EEG signal ($\mathbf{y}$), ','$\lambda_1$ = ', num2str(simdata.lam0_sapr), ...
', $\lambda_2$ = ', num2str(simdata.lam1_sapr), ', $\eta$ = ', num2str(simdata.eta_sapr),...
', $\mu$ = ', num2str(simdata.mu_sapr)];
txt_2 = [method, ', K-Complex signal ($\mathbf{x}_2$)'];
txt_3 = [method, ', Teager-Kaiser energy operator, Threshold = ', num2str(simdata.th_sapr)];
txt_4 = [method, ', Detected EEG K-complexes'];
end
N = length(y);
n = 0:N-1;
%% Plot
figure('rend','painters','pos',[100 100 550 500]);
clf
subplot(4,1,1);
text(-0.1,0.98,'(a)','Units', 'Normalized', 'VerticalAlignment', 'Top'); hold on;
plot(n/fs, y, 'k'); hold on;
p0 = plot(n/fs, x_ex3, 'r'); hold off;
title(txt_1,'interpreter','latex')
legend([p0], {'K-complex'},'location','north');
legend boxoff;
set(gca, 'box', 'off')
axis tight;
subplot(4,1,2);
text(-0.1,0.98,'(b)','Units', 'Normalized', 'VerticalAlignment', 'Top'); hold on;
p0 = plot(n/fs, kc, 'k'); hold on;
p1 = plot(n/fs, k, '--k'); hold off;
title(txt_2,'interpreter','latex')
if strcmp(method, 'SAPR')
legend([p1,p0], {'$\mathbf{\Psi} \mathbf{k}$', ...
'$\mathbf{B}^T \mathbf{B} \mathbf{\Psi} \mathbf{k}$'},'interpreter','latex', ...
'location','north');
legend boxoff;
end
set(gca, 'box', 'off')
axis tight;
subplot(4,1,3);
text(-0.1,0.98,'(c)','Units', 'Normalized', 'VerticalAlignment', 'Top'); hold on;
p0 = plot(n/fs, tk); hold on;
p1 = plot(n/fs, th*ones(1,N), 'r--'); hold off;
title(txt_3,'interpreter','latex')
legend([p0,p1], {'TKEO','Threshold'},'location','north');
legend boxoff;
set(gca, 'box', 'off')
axis tight;
subplot(4,1,4);
text(-0.1,0.98,'(d)','Units', 'Normalized', 'VerticalAlignment', 'Top'); hold on;
plot(n/fs, binX, 'Color', [0,0,0]+0.7,'linewidth',1.0); hold on;
plot(n/fs, binA, 'k');
title(txt_4,'interpreter','latex')
xlabel('Time (seconds)','interpreter','latex')
legend('Expert',method,'location','north');
legend boxoff
set(gca, 'box', 'off')
ylim([-0.2, 1.2])
printme_pdf = @(ex,meth) print('-dpdf', sprintf('../../results/%s_%s',ex,meth));
printme_pdf('ex6',lower(method));
% printme_eps = @(ex,meth) print('-depsc', sprintf('figures/%s_%s',ex,meth));
% printme_eps('ex6',lower(method));
end
程序:
数据: